来源:未知 时间:2025/02/18 作者:admin2021
这篇Analytical Chemistry文章发表于2024年12月;文章的第一作者为Junyang Chen,通讯作者为Wei Li,工作单位为郑州大学基础医学院。该研究通过针对FAP蛋白糖基化的适配体筛选策略,成功开发出一种基于双适配体的表面增强拉曼散射(SERS)免疫夹心测定法,实现了对FAP的高灵敏度和高选择性检测,为癌症诊断提供了一种新的、高效且具有临床应用潜力的检测方法。

这篇论文中的模拟与计算部分主要集中在分子对接分析,旨在揭示适配体与FAP蛋白之间的可能结合区域和相互作用机制。通过分子对接模拟,研究者能够预测适配体与FAP蛋白的结合模式,并为实验结果提供理论支持。 揭示结合机制:通过分子对接分析,研究者希望了解适配体Seq2T与FAP蛋白之间的具体结合位点和相互作用模式。 验证筛选结果:分子对接结果可以验证通过SELEX筛选得到的适配体是否能够特异性识别FAP蛋白的糖基化位点 适配体结构预测:使用Multiple Em for the Motif Elicitation(MEME)工具预测适配体的可能结构域(motif),并结合二级结构预测软件生成适配体的二级结构。 分子对接工具:使用HDOCK网络服务进行分子对接模拟。HDOCK是一种基于蛋白质-核酸相互作用的在线对接工具,能够预测核酸适配体与蛋白质之间的结合模式。 结构优化:通过将G-四链体结构纳入预测算法,进一步优化适配体的二级结构预测,使其更接近实际的结合状态。 结合位点:分子对接结果显示,适配体Seq2T与FAP蛋白的结合位点主要集中在FAP的糖基化位点附近(如ASN92和ASN314位点),这与实验结果一致。 关键残基:参与结合的关键残基包括FAP蛋白的LYS96、ASN99、LYS120、LEU121、VAL282、SER288、ARG324和THR329,以及适配体Seq2T的G22、G23、G27、C28、C43、G44、A45和G46。 结合模式:分子对接模型表明,适配体Seq2T通过其G-四链体结构与FAP蛋白的糖基化位点附近的残基形成稳定的相互作用,从而实现特异性识别。
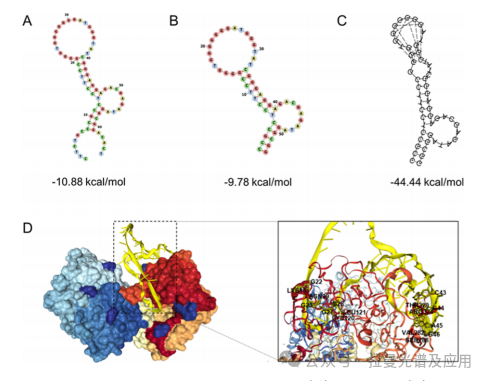 图3(D):展示了适配体Seq2T与FAP蛋白的分子对接模型,详细标注了参与结合的关键残基。图中显示了适配体Seq2T与FAP蛋白的结合位点主要集中在FAP的糖基化位点附近(ASN92和ASN314位点)。参与结合的关键残基包括FAP蛋白的LYS96、ASN99、LYS120、LEU121、VAL282、SER288、ARG324和THR329,以及适配体Seq2T的G22、G23、G27、C28、C43、G44、A45和G46。这些残基的相互作用为适配体的特异性识别提供了理论依据。
图3(D):展示了适配体Seq2T与FAP蛋白的分子对接模型,详细标注了参与结合的关键残基。图中显示了适配体Seq2T与FAP蛋白的结合位点主要集中在FAP的糖基化位点附近(ASN92和ASN314位点)。参与结合的关键残基包括FAP蛋白的LYS96、ASN99、LYS120、LEU121、VAL282、SER288、ARG324和THR329,以及适配体Seq2T的G22、G23、G27、C28、C43、G44、A45和G46。这些残基的相互作用为适配体的特异性识别提供了理论依据。
过分子对接分析,研究者不仅揭示了适配体与FAP蛋白之间的结合机制,还验证了通过SELEX筛选得到的适配体能够特异性识别FAP蛋白的糖基化位点。这些模拟结果为实验设计和适配体优化提供了重要的理论支持,有助于进一步开发基于适配体的高灵敏度和高选择性的检测方法。



